Coronavirus: la mappa delle proteine attivate dall’azione di Sars-CoV-2

Utilizzando gli strumenti della bioinformatica, un nuovo studio mette in luce i principali meccanismi di attacco del virus e quelli di difesa delle cellule umane: informazioni che potrebbero rivelarsi utili per indirizzare lo sviluppo di nuove terapie farmacologiche.
Cosa succede quando il coronavirus Sars-CoV-2 – il responsabile della pandemia di Covid-19 – entra in contatto con una cellula bronchiale umana? Un gruppo di ricercatori dell’Università di Bologna e dell’Università di Catanzaro ha messo a punto una mappa delle interazioni che avvengono tra le proteine del virus e quelle umane, mostrando quali sono “attivate” e quali “disattivate” dall’azione di Sars-CoV-2.
“Conoscere gli effetti molecolari di questo virus sulle proteine umane è fondamentale per definire strategie farmacologiche efficaci”, dice Federico M. Giorgi, ricercatore dell’Università di Bologna che ha coordinato lo studio. “Inibire le interazioni che abbiamo evidenziato potrebbe costituire una via terapeutica in grado di limitare gli effetti distruttivi del Sars-CoV-2 e di altri coronavirus sulle cellule umane”.
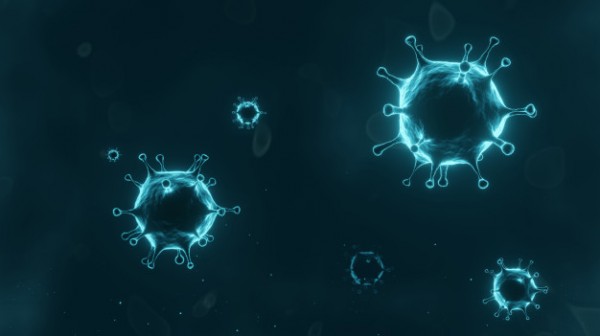
L’analisi – pubblicata sul Journal of Clinical Medicine – ha permesso di evidenziare sia meccanismi di difesa delle cellule umane, ad esempio per contrastare l’ingresso del virus, sia strategie utilizzate dal Sars-CoV-2 per diffondersi nell’organismo, ad esempio attraverso proteine che favoriscono la sua replicazione.
UN APPROCCIO INTEGRATO
I beta-coronavirus sono una sottofamiglia dei coronavirus che provoca principalmente malattie respiratorie ed intestinali. Ad oggi conosciamo sette beta-coronavirus in grado di attaccare gli umani, di cui tre particolarmente pericolosi: Sars-CoV, il virus che provoca la Sars, Mers-CoV, che provoca la Mers, e il nuovo coronavirus Sars-CoV-2, che causa la Covid-19 e ha già contagiato più di un milione di persone in tutto il mondo.
Sappiamo che Sars-CoV-2 condivide molti tratti con gli altri due beta-coronavirus “cugini”, in particolare con Sars-CoV, ma al momento non esiste ancora una descrizione dettagliata del meccanismo con cui il nuovo coronavirus attacca le cellule umane. Per fare luce su questo aspetto, utilizzando gli strumenti della bioinformatica, i ricercatori hanno combinato l’interattoma – cioè l’insieme delle interazioni tra proteine – relativo all’incontro tra Sars-CoV-2 ed una cellula umana con il patrimonio di informazioni sul comportamento a livello genetico dei virus Sars-CoV e Mers-CoV.
Per chiarire la simbologia dell’immagine, potremo dire che le proteine “disattivate” (quadrati blu) dal virus sono molte, incluse le proteine di superficie TMPRSS2 e ACE2, mentre alcune proteine umane vengono “attivate” dall’ingresso del virus (quadrati rossi), fra cui MCL1, un regolatore della morte cellulare o “apoptosi” (Immagine: Journal of Clinical Medicine).
“Questo approccio integrato, basato sulle nostre conoscenze degli altri beta-coronavirus e su quanto abbiamo scoperto finora del nuovo coronavirus, ha permesso di identificare i principali fattori che governano l’azione di Sars-CoV-2”, spiega Giorgi. “In questo modo è stato possibile generare una mappa che mostra quali proteine vengono attivate, aumentandone la produzione, e quali sono disattivate, diminuendone la quantità, quando il virus attacca una cellula del sistema respiratorio umano”.
ATTACCO E DIFESA
L’analisi degli studiosi ha permesso di identificare alcune proteine che svolgono un ruolo rilevante nel momento in cui il nuovo coronavirus entra in contatto con una cellula umana. Una di queste (chiamata MCL1) è un regolatore della morte cellulare (apoptosi) e viene attivata dalla cellula come meccanismo di difesa antivirale: per cercare di fermare l’attacco del virus, la cellula avvia una serie di reazioni che ne provocano la morte. L’azione di altre proteine viene invece ridotta al momento dell’incontro con il coronavirus. Ad esempio la proteina EEF1A1, il cui “spegnimento” riduce la capacità del virus di replicarsi.
Al tempo stesso, però, ci sono alcuni meccanismi che vengono sfruttati da Sars-CoV-2 per diffondersi nell’organismo. Tra questi, i ricercatori hanno evidenziato ad esempio la capacità del virus di ridurre l’attività dei mitocondri (gli organelli che permettono la respirazione cellulare), oppure l’azione di specifiche proteine virali (NSP7 e NSP13) capaci di disattivare alcuni meccanismi di difesa cellulare, o ancora l’aumento della produzione di proteine che favoriscono il metabolismo dell’RNA e quindi l’azione e la replicazione del virus (il cui genoma è composto, appunto, da un singolo filamento di RNA).
C’è poi la proteina ACE2, il cui ruolo nelle infezioni da beta-coronavirus è ben noto: è la proteina con cui interagiscono le “punte” del coronavirus e che permette il suo ingresso nella cellula. L’analisi mostra che le cellule si difendono dall’attacco diminuendo la presenza di ACE2. Al tempo stesso, però, i ricercatori notano che la minore presenza di questa proteina può finire per danneggiare i tessuti polmonari, favorendo così comunque la diffusione del virus.
“Tutte queste informazioni relative agli effetti del nuovo coronavirus sulle proteine delle cellule umane possono essere fondamentali per indirizzare lo sviluppo di nuove terapie farmacologiche, visto che le comuni terapie antivirali sembrano essere poco efficaci”, dice ancora Federico M. Giorgi. “Gli ultimi sviluppi in campo farmaceutico permettono infatti di sviluppare rapidamente nuove molecole, che possono rivelarsi efficaci sia per contrastare l’azione delle proteine del virus sia per rafforzare la risposta delle cellule umane”.
Infine, gli studiosi hanno analizzato la presenza di ACE2 anche per cercare di chiarire l’origine animale del coronavirus Sars-CoV-2, inizialmente connessa al pipistrello ma in seguito attribuita anche al pangolino. Dall’analisi realizzata è emersa una maggiore somiglianza tra la proteina ACE2 delle cellule umane e quelle dei pangolini: un risultato che va a sostegno dell’ipotesi secondo cui il piccolo mammifero potrebbe essere stato l’ospite originale del virus Sars-CoV-2 oppure un ospite intermedio tra il pipistrello e l’uomo.
I PROTAGONISTI DELLO STUDIO
Lo studio è stato pubblicato sul Journal of Clinical Medicine con il titolo “Master Regulator Analysis of the SARS-CoV-2/Human Interactome”. Hanno partecipato Federico M. Giorgi, Daniele Mercatelli e Carmine Ceraolo del Dipartimento di Farmacia e Biotecnologie dell’Università di Bologna, insieme a Pietro H. Guzzi dell’Università degli Studi “Magna Graecia” di Catanzaro.
MDG